生物信息學/bwa
軟體介紹
[編輯]新的DNA測序技術產生了大量的短read,需要開發快速準確的read比對程序。MAQ是一種基於哈希表的方法,用於從單個個體比對短read,但對於頻繁出現插入缺失的較長read和數百個個體的比對不合適。為了解決這些限制,開發了Burrows-Wheeler Alignment工具(BWA),它可以有效地將短測序read與大型參考序列(例如人類基因組)進行比對,允許不匹配(misMactch)和間隙(gap)。BWA支持基於鹼基空間的read,例如Illumina測序機器和來自AB SOLiD機器的顏色空間read。模擬和真實數據的評估表明,BWA比MAQ快約10-20倍,同時實現相似的準確性。此外,BWA輸出新的標準SAM(Sequence Alignment/Map)格式的比對。在比對之後進行變異分析和其他下游分析可以使用開源的SAMtools軟體包。
軟體網址:http://maq.sourceforge.net。
對參考基因組進行短讀比對方面已經發展了很多算法,但是對於長讀(>200 bp)的比對,現有的算法大多針對低測序錯誤率的短讀進行調整,不適用或者效率不高。因此,需要設計一種能夠高效比對長序列的算法。作者基於Burrows-Wheeler算法提出了一種新算法,稱為Burrows-Wheeler Aligner's Smith-Waterman Alignment(BWA-SW),能夠使用少量內存對長達1 Mb的序列進行比對。結果顯示,該算法比BLAT更加準確,與SSAHA2的準確率相當,並且比兩者都快幾十倍。BWA-SW的原始碼已公開。
bwa-sw軟體網站:http://bio-bwa.sourceforge.net/
軟體發表時的文章:
使用方法
[編輯]本頁面主要介紹BWA的原理,關於使用請參考BWA的使用。
背景介紹
[編輯]bwa軟體開發的背景
[編輯]將Illumina/Solexa測序技術產生的短read序列比對到大型基因組的比對軟體,分為三個類別:基於哈希的比對程序、基於基因組的比對程序和基於字符串合併排序的比對程序。最近,使用Burrows-Wheeler Transform(BWT)的字符串匹配理論引起了幾個研究小組的關注,這導致了SOAPv2(http://soap.genomics.org.cn/)、Bowtie(Langmead et al., 2009)和BWA等比對軟體的開發。BWA是一種新的比對程序,使用BWT和反向搜索(Ferragina 和 Manzini,2000 年;Lippert,2005 年)來進行精確和近似匹配,具有相對較小的內存占用(Lam 等人,2008 年),能夠快速且準確地將短read序列比對到大型基因組。BWA比對軟體被證明在模擬和真實數據上的表現優於其他比對程序,特別是在內存使用方面。
- 基於哈希的比對程序:Eland (Cox, 2007, unpublished material)、RMAP (Smith et al., 2008)、MAQ (Li et al., 2008a)、ZOOM (Lin et al., 2008)、SeqMap (Jiang and Wong, 2008)、CloudBurst (Schatz, 2009) 和 SHRiMP (http://compbio.cs.toronto.edu/shrimp)
- 基於基因組的比對程序:SOAPv1 (Li et al., 2008b)、PASS (Campagna et al., 2009)、MOM (Eaves and Gao, 2009)、ProbeMatch (Jung Kim et al., 2009)、NovoAlign (http://www.novocraft.com)、ReSEQ (http://code.google.com/p/re-seq)、Mosaik (http://bioinformatics.bc.edu/marthlab/Mosaik) 和 BFAST (http://genome.ucla.edu/bfast)
- 基於字符串合併排序:Malhis 等人,2009 年
BWA-SW算法
[編輯]隨著 1990 年左右的局部比對軟體的發展,例如 FASTA(Pearson 和 Lipman,1988)和 BLAST(Altschul 等,1997),自 2000 年以來開發了新一代更快的 DNA 序列匹配方法,包括 MegaBLAST (Morgulis et al., 2008; Zhang et al., 2000)、SSAHA2 (Ning et al., 2001)、BLAT (Kent, 2002) 和 PatternHunter (Ma et al., 2002),大大加快了匹配測序read針對大型參考基因組。當新的測序技術出現並產生數百萬個短read(<100 bp)時,各種新算法被開發出來,速度提高了 10-1000 倍,包括 SOAP (Li,R. et al., 2008)、MAQ (Li, H. et al., 2008)、 Bowtie(Langmead 等人,2009 年)和 BWA(Li 和 Durbin,2009 年)。然而,Roche/454 測序技術已經開始產生了 >400 bp 的read讀數,Illumina 逐漸增加了 >100 bp 的read讀數,而 Pacific Bioscience 在早期測試中產生了 1000 bp 的讀數(Eid 等,2009)。來自新測序技術的read不再是短的序列,這有效地排除了許多專為不超過 100 bp 的read設計的比對軟體。
隨著產生長read讀數的新測序技術的出現,現有的短read讀數比對算法對於長read讀數比對來說並不高效。長read比對的目的是尋找局部匹配,因為長read更容易受到結構變異和參考基因組序列中錯配組裝的影響,但受靠近read末端的錯配影響較小。此外,由於長read中更頻繁地出現插入和缺失,所以長讀數比對算法必須對比對缺口gap持寬容態度。
BWA-SW是一種新的長read排列算法,在兩個FM-indices之間使用動態編程來尋找種子,並在它們在參考序列中很少出現的情況下進行擴展。該算法旨在減少高度重複序列的不必要的擴展,從而提高速度。
BWA原理方法
[編輯]前綴樹和字符串匹配
[編輯]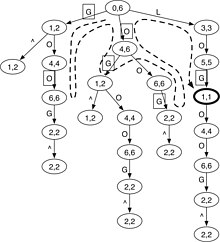
字符串 X 的前綴樹是一棵樹,其中每條邊都用一個符號標記,從葉子到根的路徑上邊符號的字符串連接給出了唯一的前綴 X。在前綴樹上,字符串連接從節點到根的邊符號給出了 X 的唯一子串,稱為節點所代表的串。請注意,X 的前綴樹與 X 的反向後綴樹相同,因此後綴樹理論也可以應用於前綴樹。
使用前綴 trie,測試查詢 W 是否是 X 的精確子串相當於找到表示 W 的節點,這可以通過將 W 中的每個符號與一條邊匹配,在 O(|W|) 時間內完成,從根。為了允許不匹配,可以徹底遍歷特里樹並將 W 匹配到每個可能的路徑。稍後將展示如何通過使用 W 的前綴信息來加速此搜索。圖1給出了「GOOGOL」的前綴樹的示例。每個節點中的後綴數組(SA)間隔在第 2.3.3 節中解釋。
Burrows–Wheeler轉換
[編輯]
設 Σ 是一個字母表。 符號 $ 不存在於 Σ 中,並且按字典順序小於 Σ 中的所有符號。 字符串 X=a0a1…an−1 總是以符號 $ 結尾(即 an−1=$),並且這個符號只出現在末尾。 設 X[i]=ai, i=0, 1,..., n−1, 是 X 的第 i 個符號,X[i, j]=ai ...aj 是一個子串,Xi=X[i, n− 1] X 的後綴。X 的後綴數組 S 是整數 0…n−1 的排列,使得 S(i) 是第 i 個最小後綴的起始位置。 X 的 BWT 在 S(i)=0 時定義為 B[i]=$,否則定義為 B[i]=X[S(i)-1]。 還將字符串 X 的長度定義為 |X| 因此 |X|=|B|=n。 圖2 給出了如何構造 BWT 和後綴數組的示例。
圖 2 所示的算法在時間和空間上是二次的。 然而,這不是必需的。 在實踐中,通常先構造後綴數組,然後生成 BWT。 大多數構建後綴數組的算法至少需要 n⌈log2n⌉ 位的工作空間,對於人類基因組來說,這相當於 12 GB。 最近,Hon 等人。 (2007) 給出了一種新算法,該算法使用 n 位工作空間,並且在構建人類基因組 BWT 的尖峰時間只需要 <1 GB 內存。 該算法在 BWT-SW 中實現(Lam 等人,2008 年)。 修改了原始碼以使其與 BWA 一起使用。
後綴數組間隔和序列比對
[編輯]如果字符串 W 是 X 的子字符串,則 X 中 W 每次出現的位置將出現在後綴數組中的一個區間內。 這是因為所有以 W 作為前綴的後綴都排序在一起。 基於這一觀察,定義:
(1)
(2)
特別是,如果 W 是空字符串,則 R(W)=1 和。
稱為 W 的 SA 區間,X 中所有出現 W 的位置集合是。 例如在圖 2 中,字符串 『go』 的 SA 區間為 [1, 2]。 此區間中的後綴數組值為 3 和 0,給出了所有「go」出現的位置。
知道後綴數組中的間隔可以得到位置。 因此,序列比對相當於搜索X的子串的SA區間與查詢匹配。 對於精確匹配問題,只能找到一個這樣的區間; 對於不精確匹配的問題,可能有很多。
精確匹配:向後搜索
[編輯]令 C(a) 是 C(a)=#{0≤j≤n−2:X[j]<a}和O(a,i)=#{0≤j≤i:B[j]<a}中按字典順序小於 a ∈ Σ 的符號數,O(a, i) 是 a 在 B[0, i] 中的出現次數。 Ferragina 和 Manzini (2000) 證明了如果 W 是 X 的子串:
(3)
(4)
若且唯若 aW 是 X 的子串時,。此結果使得可以通過迭代計算 和 來測試 W 是否是 X 的子串並計算 W 在 O(|W|) 時間內的出現次數從W結尾。這個過程叫做反向搜索。
需要注意的是,等式(3)和(4)實際上實現了對X的前綴trie的自頂向下遍歷,因為如果知道其父節點的間隔,可以在恆定時間內計算出子節點的SA間隔 . 從這個意義上說,向後搜索相當於前綴樹上的精確字符串匹配,但沒有明確地將樹放入內存中。
不精確匹配:有界遍歷/回溯
[編輯]給出了一種遞歸算法,用於搜索 X 的子串的 SA 區間,該區間與查詢字符串 W 匹配且差異不超過 z(不匹配或間隙)。 本質上,該算法使用向後搜索從基因組中採樣不同的子串。 此過程受 D(·) 數組的限制,其中 D(i) 是 W[0, i] 中差異數量的下限。 D 估計得越好,搜索空間越小,算法效率越高。 通過為所有 i 設置 D(i)=0 來實現一個樸素的界限,但由此產生的算法在差異數量上顯然是指數級的,並且效率較低。
Precalculation:
Calculate BWT string B for reference string X
Calculate array C(·) and Ο(·,·) from B
Calculate BWT string B' for the reverse reference
Calculate array Ο'(·) from B'
Procedures:
INEXACTSEARCH(W,z)
CalculateD(W)
return InexRecur(W, |W|-1,z,1,|X|-1)
CalculateD(W)
k ← 1
z ← 0
for i = 0 to |W|-1 do
k ← C(W[i])+O'(W[i],k-1)+1
l ← C(W[i])+O'(W[i],l)
if k > l then
k ← l
l ← |X|-1
z ← z+1
D(i) ← z
InexRecur( W, z, k, l)
if z < D(i) then
return Φ
if z <0 then
return {[k,l]}
I ← Φ
I ← I ∪ InexRecur(W, i-1,z-1,k,l)
for each b∈{A,C,G,T} do
k ← C(b) + O(b,k-1)+1
l ← C(b)+O(b,l)
if k ≤ l then
I ← I ∪ InexRecur(W,i,z-1,k,l)
if b = W[i] then
I ← I ∪ InexRecur(W,i- 1 ,z,k,l)
else
I ← I ∪ InexRecur(W,i-1 ,z-1,k,l)
return I
圖3:不精確搜索匹配 W 的子串的 SA 區間的算法。參考 X 以 $ 終止,而 W 以 A/C/G/T 終止。 過程 InexactSearch(W, z) 返回與 W 匹配的子串的 SA 間隔,差異不超過 z(不匹配或間隙); InexRecur(W, i, z, k, l) 在後綴Wi+1匹配區間[k, l]的條件下,遞歸計算匹配W[0, i]且差異不超過z的子串的SA區間。 以星號開頭的行分別用於插入和刪除 X。 D(i) 是字符串 W[0, i] 中差異數量的下限。圖 3 中的 CalculateD 過程提供了一個更好但不是最佳的界限。 它在概念上與圖 4 中描述的相同,後者更易於理解。 使用反向(未補碼)參考序列的 BWT 來測試 W 的子串是否也是 X 的子串。請注意,單獨使用 BWT 字符串 B 進行此測試將使CalculateD 成為O(|W|2) 過程 ,而不是圖 3 中描述的 。
CalculateD(W)
z ← 0
j ← 0
for i=0 to |W| — 1 do
if W[j,i] is not a substring of X then
z ← z+1
j ← i+l
D(i) ← z
圖4:計算 D(i) 的等效算法。
為了理解D的作用,回到在X=GOOGOL$中搜索W=LOL的例子(圖1)。如果為所有 i 設置 D(i)=0 並禁止間隙(去除算法中的兩條星線),則 InexRecur 的調用圖是一棵樹,有效地模仿了圖 1 中虛線所示的搜索路線. 然而,通過CalculateD,知道D(0)=0 和D(1)=D(2)=1。然後可以避免下降到前綴樹中的「G」和「O」子樹以獲得更小的搜索空間。
圖 3 中的算法保證找到所有允許最大 z 差異的區間。理論上是完整的,但在實踐中,也做了各種修改。首先,對不匹配、間隙開放和間隙擴展支付不同的懲罰,這對生物數據更現實。其次,使用類似堆的資料結構來保持部分命中而不是使用遞歸。堆狀結構優先於部分命中的對齊分數,以使 BWA 始終首先找到最佳間隔。同時處理反向互補讀取序列。請注意,圖 3 中描述的遞歸有效地模擬了前綴樹上的深度優先搜索 (DFS),而 BWA 使用這種類似堆的資料結構實現了廣度優先搜索 (BFS)。第三,採用迭代策略:如果頂部區間是重複的,默認不搜索次優區間;如果頂部間隔是唯一的並且具有 z 差異,則只搜索最多具有 z + 1 差異的命中。這種迭代策略在保持生成映射質量的能力的同時加速了 BWA。然而,這也使得 BWA 的速度對讀取和參考之間的不匹配率敏感,因為找到具有更多差異的命中通常更慢。第四,允許對讀取的前幾十個鹼基對的最大允許差異設置限制,稱之為種子序列。給定 70 bp 模擬讀數,與 32 bp 種子中最大兩個差異的比對比沒有種子快 2.5 倍。對齊錯誤率,即模擬中置信映射中錯誤對齊的比例(另見 第 2.3.3 節),僅從 0.08% 增加到 0.11%。對於較短的讀取,播種效果較差。
降低內存
[編輯]上述算法需要在內存中加載出現數組O和後綴數組S。保存完整的 O 和 S 數組需要巨大的內存。幸運的是,可以通過只存儲 O 和 S 數組的一小部分並動態計算其餘部分來減少內存。 BWT-SW(Lam 等人,2008 年)和 Bowtie(Langmead 等人,2009 年)使用了由 Ferragina 和 Manzini(2000 年)首次引入的類似策略。
給定大小為 n 的基因組,出現數組 O(·, ·) 需要 位,因為每個整數需要 位,並且數組中有 4n 個。在實踐中,在內存中存儲 O(·, k) 的 k 為 128 的因子,並使用 BWT 字符串 B 計算其餘元素。當使用兩位表示核苷酸時,B 需要 2n 位。因此,用於反向搜索的內存為 2n+n⌈log2n⌉/32 位。由於還需要存儲反向基因組的 BWT 來計算邊界,因此計算間隔所需的內存增加了一倍,或者對於 3 Gb 基因組大約需要 2.3 GB。
枚舉每個出現的位置需要後綴數組 S。如果將整個 S 放在內存中,它將使用 n⌈log2n⌉ 位。然而,當知道其中的一部分時,也可以重建整個 S。事實上,S 和逆壓縮後綴數組 (inverse CSA) (Grossi and Vitter, 2000) 滿足:
(5)
其中 表示將變換 重複應用 j 次。 逆 CSA 可以用出現數組 O 計算:
(6)
在 BWA 中,只在內存 S(k) 中存儲可以被 32 整除的 k。對於不是 32 的因數的 k,重複應用 直到對於某些 j, 是 32 的因數,然後可以查找 並且可以使用等式 (5) 計算 S(k)。
總之,比對過程使用 4n+n⌈log2n⌉/8 位,或 n 個字節用於小於 4 Gb 的基因組。 這包括原始和反向基因組的 BWT 字符串、部分出現數組和部分後綴數組的內存。 此外,堆、緩存和其他資料結構需要幾百兆字節的內存。
Illumina read的其他實際問題
[編輯]不明確的鹼基
[編輯]read上的非 A/C/G/T 鹼基被簡單地視為不匹配,這在算法中是隱含的(圖 3)。參考基因組上的非 A/C/G/T 鹼基被轉換為隨機核苷酸。這樣做可能會導致錯誤命中充滿模糊鹼基的區域。幸運的是,鑑於相對較長的讀取時間,發生這種情況的可能性非常小。嘗試了 200 萬個 32 bp 的讀數,但沒有看到任何讀數偶然映射到 poly-N 區域。
雙端映射
[編輯]BWA 支持雙端映射。它首先找到所有好的命中的位置,根據染色體坐標對它們進行排序,然後對所有潛在的命中進行線性掃描以配對兩端。計算所有染色體坐標需要經常查找後綴數組。這種配對過程非常耗時,因為使用上述方法即時生成完整的後綴數組非常昂貴。為了加速配對,緩存了大的間隔。此策略將花費在配對上的時間減半。
在配對中,BWA 批量處理 256K 讀取對。在每個批次中,BWA 將完整的 BWA 索引加載到內存中,為每次出現生成染色體坐標,從兩端映射的映射質量高於 20 的讀取對估計插入大小分布,然後將它們配對。之後,BWA 從內存中清除 BWT 索引,加載 2 位編碼參考序列,並對未映射的讀取執行 Smith-Waterman 對齊,其配偶可以可靠地對齊。 Smith-Waterman 對齊挽救了一些差異過大的讀取。
確定允許的最大差異數
[編輯]給定長度為 m 的讀取,BWA 僅容忍最多具有 k 個差異(錯配或間隙)的命中,其中選擇 k 使得 <4% 的 m 長讀取和 2% 的統一鹼基錯誤率可能包含大於 k 的差異.使用此配置,對於 15–37 bp 讀取,k 等於 2;對於 38–63 bp,k=3;對於 64–92 bp,k=4;對於 93–123 bp,k=5;對於 124–156 bp 讀數,k=6。
生成映射質量分數
[編輯]對於每個比對,BWA 計算一個映射質量分數,這是比對不正確的 Phred 標度機率。該算法與 MAQ 相似,不同之處在於在 BWA 中假設總能找到真正的命中。進行此修改是因為知道 MAQ 的公式高估了錯過真正命中的機率,從而導致低估了映射質量。模擬表明,由於這種修改,BWA 可能會高估映射質量,但偏差相對較小。例如,BWA 在映射質量為 60 的 1 569 108 個模擬 70 bp 讀數中錯誤地對齊了 11 個讀數。這些 Q60 映射的錯誤率 7 × 10−6 (= 11/1 569 108) 高於理論預期 10-6。
SOLiD read的比對
[編輯]對於 SOLiD 讀取,BWA 將參考基因組轉換為二核苷酸「顏色」序列,並為顏色基因組構建 BWT 索引。讀取被映射在顏色空間中,其中序列的反向補碼與反向相同,因為顏色的補碼是它本身。對於SOLiD配對末端作圖,如果兩種情況中的任何一種為真,則稱讀數對處於正確的方向:(i)兩端都映射到基因組的正向鏈,其中R3讀數具有較小的坐標; (ii) 兩端映射到基因組的反向鏈,F3 讀數具有較小的坐標。 Smith-Waterman 對齊也在色彩空間中完成。
比對後,BWA 使用動態編程將顏色讀取序列解碼為核苷酸序列。給定核苷酸參考子序列 和映射到該子序列的顏色讀取序列 ,BWA 推斷出核苷酸序列 。
其中 q' 是突變的 Phred 標度機率,qi 是顏色 ci 的 Phred 質量,函數 g(b, b')=g(b', b) 給出對應於兩個相鄰核苷酸 b 和 b' 的顏色。本質上,如果,將支付懲罰 q′。和一個懲罰 qi 如果。
這種優化可以通過動態編程來完成,因為超出位置 i 的最佳解碼僅取決於選擇。 Let 是達到 i 的最佳解碼分數。 迭代方程為
BWA 近似基本質量如下。 Let 一個包含圖片、插圖等的外部文件。
對象名稱是 btp324i11.jpg。 第 i 個基本質量 ,i=2…l,計算如下:
BWA 輸出序列,質量為 作為 SOLiD 映射的最終結果。
結果
[編輯]說明
[編輯]基於參考基因組的 BWT 實現了 BWA 來進行短讀比對。 為單端讀取執行缺口比對,支持雙端比對,生成比對質量並在需要時提供多次命中。 默認的輸出對齊格式是 SAM(序列比對/映射格式)。 可以使用 SAMtools (http://samtools.sourceforge.net) 來提取區域中的比對、合併/排序比對、獲得單核苷酸多態性 (SNP) 和 indel 調用並可視化比對。
文檔和原始碼可在 MAQ 網站免費獲得:http://maq.sourceforge.net。
評估
[編輯]為了評估 BWA 的性能,測試了另外三個比對程序:MAQ(Li 等人,2008a)、SOAPv2(http://soap.genomics.org.cn)和 Bowtie(Langmead 等人,2009 年)。 MAQ 索引使用哈希表read並掃描基因組,是之前為大規模read比對開發的軟體包。 SOAPv2 和 Bowtie 是另外兩個基於 BWT 的短讀比對軟體。最新的 SOAP-2.1.7(Li 等人,未公開數據)使用 2way-BWT(Lam 等人,未公開數據)進行對齊。可以接收超過 35 bp 種子序列的更多錯配,並支持僅限於一個缺口開放的缺口比對。 Bowtie(版本 0.9.9.2)部署了與 BWA 類似的算法,並沒有通過用 D(i) 限制搜索來減少搜索空間,而是通過巧妙地對原始和反向讀取序列進行對齊以繞過對前綴樹的根進行不必要的搜索。默認情況下,Bowtie 對前綴 trie 執行 DFS,並在找到第一個符合條件的命中時停止。因此,即使其播種策略被禁用,它也可能會錯過最佳的不精確命中。可以通過在命令行應用「--best」來使 Bowtie 執行 BFS,但這會使 Bowtie 變慢。
包括 BWA 在內的所有四個程序都會在多個同樣最佳的位置上隨機放置重複讀取。由於主要對實踐中的置信比對感興趣,因此需要排除重複命中。 SOAPv2 給出讀取的同等最佳命中數。僅保留唯一映射。還要求 SOAPv2 將可能的間隙大小限制為最多 3 bp。使用命令行選項「--best -k 2」運行 Bowtie,使 Bowtie 輸出讀取的前兩個命中。如果第二個最佳命中包含與最佳命中相同數量的不匹配,則丟棄讀取對齊。 MAQ 和 BWA 生成映射質量。使用 MAQ 的比對質量閾值 1 和 BWA 的 10 來確定可信映射。使用不同的閾值是因為知道 MAQ 的映射質量被低估了,而 BWA 的映射質量被高估了。
模擬數據評估
[編輯]使用 SAMtools 包中包含的 wgsim 程序模擬來自人類基因組的讀數,並運行四個程序將讀數比對回人類基因組。 由於知道每個read的確切坐標,能夠計算比對錯誤率。
表 1 顯示 BWA 和 MAQ 實現了相似的比對精度。 在可信映射讀取的比例和可信映射的錯誤率方面,BWA 比 Bowtie 和 SOAPv2 更準確。 請注意,SOAP-2.1.7 針對長度超過 35 bp 的讀取進行了優化。 對於 32 bp 讀取,SOAP-2.0.1 優於最新版本。
| Single-end | Paired-end | |||||
|---|---|---|---|---|---|---|
| Program | Time (s) | Conf (%) | Err (%) | Time (s) | Conf (%) | Err (%) |
| Bowtie-32 | 1271 | 79.0 | 0.76 | 1391 | 85.7 | 0.57 |
| BWA-32 | 823 | 80.6 | 0.30 | 1224 | 89.6 | 0.32 |
| MAQ-32 | 19797 | 81.0 | 0.14 | 21589 | 87.2 | 0.07 |
| SOAP2-32 | 256 | 78.6 | 1.16 | 1909 | 86.8 | 0.78 |
| Bowtie-70 | 1726 | 86.3 | 0.20 | 1580 | 90.7 | 0.43 |
| BWA-70 | 1599 | 90.7 | 0.12 | 1619 | 96.2 | 0.11 |
| MAQ-70 | 17928 | 91.0 | 0.13 | 19046 | 94.6 | 0.05 |
| SOAP2-70 | 317 | 90.3 | 0.39 | 708 | 94.5 | 0.34 |
| bowtie-125 | 1966 | 88.0 | 0.07 | 1701 | 91.0 | 0.37 |
| BWA-125 | 3021 | 93.0 | 0.05 | 3059 | 97.6 | 0.04 |
| MAQ-125 | 17506 | 92.7 | 0.08 | 19388 | 96.3 | 0.02 |
| SOAP2-125 | 555 | 91.5 | 0.17 | 1187 | 90.8 | 0.14 |
| 分別從人類基因組模擬了 100 萬對 32、70 和 125 bp 讀數,SNP 突變率為 0.09%,indel 突變率為 0.01%,均勻測序鹼基錯誤率為 2%。 32 bp 讀數的插入大小來自常態分佈 N(170, 25),70 和 125 bp 讀數來自 N(500, 50)。 表中顯示了 2.5 GHz Xeon E5420 處理器單核上的 CPU 時間(時間)、置信映射讀取百分比 (Conf) 和置信映射錯誤對齊百分比 (Err)。 | ||||||
在速度上,SOAPv2 是最快的,如果禁用間隙對齊,雙端映射實際上會快 30-80%。帶有默認選項(數據未顯示)的 Bowtie 比單端映射上的當前設置「–best -k 2」快幾倍。但是,速度的提高是以犧牲準確性為代價的。例如,使用默認選項,Bowtie 可以在 151 秒內映射 200 萬個單端 32 bp 讀數,但 6.4% 的置信映射是錯誤的。這種高對齊錯誤率可能會使結構變異的檢測複雜化,並可能影響 SNP 的準確性。在 BWA 和 MAQ 之間,BWA 快 6–18 倍,具體取決於讀取長度。 MAQ 的速度不受讀取長度的影響,因為它在內部將所有讀取視為 128 bp。通過不檢查類似於 Bowtie 和 SOAPv2 所做的次優命中,可以加速 BWA。然而,在這種情況下,計算映射質量是不可能的,相信生成適當的映射質量對各種下游分析有用,例如檢測結構變化。
在內存上,SOAPv2 使用 5.4 GB。 Bowtie 和 BWA 都使用 2.3 GB 用於單端映射,大約 3 GB 用於雙端映射,比 MAQ 的內存占用 1 GB 大。然而,所有三個基於 BWT 的對齊器的內存使用與要對齊的讀取數量無關,而 MAQ 在其中是線性的。此外,所有基於 BWT 的對齊器都支持多執行緒,這減少了多核計算機上每個 CPU 核心的內存。在現代計算機伺服器上,內存不是基於 BWT 的對齊器的實際問題。
真實數據評估
[編輯]為了評估真實數據的性能,從歐洲讀取檔案 (AC:ERR000589) 下載了大約 1220 萬對 51 bp 讀取。 這些讀數由 Illumina 為 NA12750 生成,NA12750 是 1000 基因組計劃 (http://www.1000genomes.org) 中的一個男性。 讀數被映射到人類基因組 NCBI build 36。表 2 顯示幾乎所有來自 MAQ 和 BWA 的可信比對都以一致的對存在,儘管 MAQ 給出的可信比對較少。 較慢的 BWA 模式(無種子;即使最高命中是重複也搜索次優命中)效果更好。 在該模式下,BWA 在 6.3 小時內自信地映射了所有讀數的 89.2%,並以一致對的方式準確映射了 99.2%。
| Program | Time (h) | Conf (%) | Paired (%) |
|---|---|---|---|
| Bowtie | 5.2 | 84.4 | 96.3 |
| BWA | 4.0 | 88.9 | 98.8 |
| MAQ | 94.9 | 86.1 | 98.7 |
| SOAP2 | 3.4 | 88.3 | 97.5 |
| 1220 萬個讀取對被對到人類基因組。 2.5 GHz 至強 E5420 處理器單核上的 CPU 時間(時間)、置信度映射讀取百分比 (Conf) 和以正確方向映射且在 300 bp 範圍內的配對置信度映射(配對),顯示在 桌子。 | |||
在這個實驗中,如果間隙對齊被禁用,SOAPv2 的速度將提高兩倍,置信度映射 (Conf) 和配對百分比 (Paired) 均下降 1%。相比之下,BWA 僅執行無間隙對齊時的速度是它的 1.4 倍。但即使禁用了基於 BWT 的缺口比對,BWA 仍然能夠通過 Smith-Waterman 比對恢復許多短插入缺失,給出配對末端讀數。
還獲得了雞基因組序列(2.1 版),並將這 1220 萬個讀取對與人雞雜交參考序列進行比對。與純人類對齊相比,置信度映射百分比幾乎沒有變化。至於映射到雞基因組的讀取數,Bowtie 將 2640、BWA 2942、MAQ 3005 和 SOAPv2 映射到錯誤的基因組的讀取數為 4531。如果考慮雞序列占人雞雜交參考的四分之一,則 BWA 的比對錯誤率約為 0.06%(=2942×4/12.2M/0.889)。請注意,這種比對錯誤率的估計可能會被低估,因為錯誤比對的人類read往往與人類的重複序列相關,並比對回人類序列。由於存在高度保守的序列和人類組裝不完整或雞基因組組裝錯誤,估計也可能被高估。
如果想要更少的錯誤,BWA 和 MAQ 生成的映射質量允許選擇精度更高的對齊。如果將確定 BWA 的置信命中的比對質量閾值提高到 25,則 86.4% 的讀數可以與比對到雞基因組的 1927 個讀數可靠地對齊,在百分比置信映射和比對到的讀數數量方面均優於 Bowtie錯誤的基因組。
討論
[編輯]對於與人類參考基因組的短讀比對,BWA 比 MAQ 快一個數量級,同時實現相似的比對精度。它支持單端讀取的間隙對齊,當讀取變得更長並且傾向於包含插入缺失時,這一點變得越來越重要。 BWA 以 SAM 格式輸出對齊,以利用 SAMtools 中實施的下游分析。 BWA 加 SAMtools 提供了 MAQ 包的大部分功能和附加功能。
與速度、內存和比對讀取的數量相比,在真實數據上評估對齊精度要困難得。本文使用了三個標準來評估比對軟體的準確性。第一個標準只能用模擬數據進行評估,它是置信比對數量和置信比對中比對錯誤率的組合。請注意,僅可信比對的數量可能不是一個好的標準:可以以犧牲準確性為代價比對更多。第二個標準是比對read的數量和比對成一致對的read數量的組合,它適用於真實數據,假設來自實驗的交配信息是正確的,並且結構變異很少。儘管此標準與比對軟體定義配對「一致性」的方式有關,但根據經驗,如果配對參數設置正確,則具有很高的信息量。第三個標準是如果有意添加來自不同物種的參考序列,read比對到錯誤參考序列的比例。
雖然理論上 BWA 適用於任意長的讀取,但它的性能在長read上會下降,尤其是在測序錯誤率很高的情況下。此外,BWA 總是需要比對完整的read,從第一個鹼基到最後一個(即相對於read的全局比對),但較長的read更有可能被參考基因組中的結構變異或錯誤組裝中斷,這將BWA 失敗。對於長讀段,可能更好的解決方案是將讀段分成多個短片段,用上述算法分別比對片段,然後加入部分比對以獲得讀段的完整比對。
BWA-SW方法
[編輯]BWA-SW算法概述
[編輯]BWA-SW 為參考序列和查詢序列構建 FM 索引。它隱含地表示前綴樹中的參考序列,並表示前綴有向無環詞圖(前綴 DAWG;Blumer 等人,1985)中的查詢序列,該圖是從查詢序列的前綴樹轉換而來的(算法圖)。通過分別遍歷參考前綴 trie 和查詢 DAWG,可以在 trie 和 DAWG 之間應用動態編程。如果不應用啟發式算法,這種動態編程將找到所有本地匹配項,但不會比 BWT-SW 快。在 BWA-SW 中,應用了兩個啟發式規則來大大加速這個過程。首先,查詢 DAWG 上的遍歷是在外循環中進行的,因此在不完成動態規劃的情況下,知道參考前綴樹中所有與查詢節點匹配且得分為正的節點。基於對真實對齊往往具有高對齊分數的觀察,可以在每個節點上修剪低分數匹配以限制僅圍繞良好匹配的動態規劃。動態規劃的規模因此可以顯著減少。在這個過程中可能會修剪真正的比對,但實際上,這可以通過使用啟發式方法來控制,並且很少發生,因為給定長或高質量的查詢序列。其次,BWA-SW 只報告在查詢序列上基本上不重疊的比對,而不是給出所有重要的局部比對。它啟發式地識別並丟棄包含在較長對齊中的種子,從而節省不成功種子擴展的計算時間。
符號和定義
[編輯]後綴數組和 BWT
[編輯]設 Σ={A, C, G, T} 是核苷酸字母表,$ 是一個在字典上比 Σ 中的所有符號都小的符號。 給定一個核苷酸序列 X=a1…an−1 和 an−1=$,讓 X[i]=ai 是第 i 個符號,X[i, j]=ai…aj 是 X 和 Xi=X 的子序列 [i, n−1] X 的後綴。 X 的後綴數組 S 是整數 0,..., n−1 的排列,使得 S(i)=j 若且唯若 Xj 是第 i 個字典序最小的 後綴。 X 的 BWT 是 X 的排列,如果 S(i)=0,則 B[i]=$,否則 B[i]=X[S(i)−1]。
後綴數組間隔
[編輯]給定一個序列 W,後綴數組 interval 或 SA interval 的 W 定義為
調頻指數
[編輯]後綴數組 S、數組 C 和 O 足以在 X 中精確搜索模式。 FM-index (Ferragina and Manzini, 2000) 是三個數組的壓縮表示,由壓縮的 BWT 字符串 B、計算 O 和後綴數組 S 的一部分。然而,BWA-SW 使用簡化的 FM 索引,其中不壓縮 B 並在沒有輔助資料結構的情況下存儲部分出現數組 O。簡化版本對於字母表非常小的 DNA 序列更有效。之前的論文 (Li and Durbin, 2009) 中介紹了有關構造的詳細信息。
比對
[編輯]對齊是一個元組 (W1, W2, A),其中 W1 和 W2 是兩個字符串,A 是一系列將 W2 轉換為 W1 的複製、替換、插入和刪除操作。插入和刪除是間隙。差距和替代是差異。對齊的編輯距離等於 A 中差異的總數。
可以為給定評分系統的比對計算分數。如果 W1 和 W2 可以與正分數對齊,說 W1 匹配 W2,在這種情況下,也說 (W1, W2) 是匹配的。
如果 W1 是 W'1 的子串,則稱匹配 (W1, W2) 包含在第一個序列的 (W'1, W'2) 中。同樣,可以定義比對(更強的條件)之間以及比對和匹配之間的「包含」關係。
前綴樹和前綴 DAWG
[編輯]
字符串 X 的前綴 trie 是一棵樹,每條邊都標有一個符號,這樣從葉子到根的路徑上的符號串聯給出了唯一的 X 前綴。 從節點到根的邊符號串聯為 總是 X 的子串,稱為節點所代表的串。 節點的SA區間定義為該節點所代表的字符串的SA區間。 不同的節點可能有相同的區間,但是回顧SA區間的定義,知道這些節點所代表的字符串一定是同一個字符串的前綴,長度不同。
X 的前綴 DAWG 通過摺疊具有相同間隔的節點從前綴 trie 轉換而來。 因此,在前綴 DAWG 中,節點和 SA 區間具有一一對應的關係,並且一個節點可能代表 X 的多個子串,落入序列中,其中每個子串都是下一個的前綴,如前一段所述。 圖 1 給出了一個例子。
將前綴樹與前綴 DAWG 對齊
[編輯]為查詢序列 W 構建了一個前綴 DAWG 𝒢(W),為參考 X 構建了一個前綴 trie 𝒯(X)。用於計算 W 和 X 之間的最佳分數的動態規劃如下。 當u是𝒢(W)的根,v是𝒯(X)的根時,設Guv=Iuv=Duv=0。 在 𝒢(W) 中的節點 u,對於它的每個父節點 u′,計算
其中 v′ 是 𝒯(X) 中 v 的父級,函數 S(u′, u; v′, v) 給出邊上的符號 (u′, u) 和邊上的符號 (v′, v) 和 q 和 r 分別是間隙開放和間隙擴展懲罰。 Guv、Iuv 和 Duv 的計算公式為:
其中 pre(u) 是 u 的父節點集。 Guv 等於 u 表示的(可能是多個)子串與 v 表示的(一個)子串之間的最佳分數。如果 Guv>0,說節點 v 與 u 匹配。
動態規劃是通過以反向後序(即所有父節點在子節點之前訪問)以嵌套方式遍歷𝒢(W) 和 𝒯(X) 來執行的。 注意到一旦u不匹配v,u不匹配v的任何節點,只需要訪問靠近𝒯(X)根節點的節點,無需遍歷整個trie,相比之下大大減少了迭代次數 到標準的 Smith-Waterman 算法,該算法總是通過整個參考序列。
標準 Smith-Waterman 的加速
[編輯]與時間複雜度為 O(|X|·|W|) 的標準 Smith-Waterman 對齊相比,BWA-SW 具有更好的時間複雜度,因為它並不比時間複雜度為 O(|X|0.628|W|) 的 BWT-SW 慢。(Lam 等人,2008 年)。這個結論是因為對於短序列比對,正在考慮多個可能的匹配與單個 uv 比較。然而,由於與遍歷前綴 trie 和前綴 DAWG 相關的複雜步驟,與每次迭代相關的常數要大得多,這使得當使用 BWA-SW 擴展唯一對齊時 BWA-SW 效率低下。更有效的策略是使用 BWA-SW 查找部分匹配並應用 Smith-Waterman 算法進行擴展。在動態規劃中,知道在任何對中考慮的部分匹配的數量,因為這可以從 SA 間隔的大小計算。當Guv足夠好並且v的SA區間大小低於某個閾值(默認為3)時,保存(u,v)對,稱為種子區間對,不要從𝒯中的v節點往更深處(X)。通過查找 X 和 W 的後綴數組,可以從種子間隔對中導出種子匹配,或簡稱為種子。這些種子隨後由 Smith-Waterman 算法擴展。如果整個查詢是一個高度重複的序列,它將完全按照上一節中描述的算法進行對齊,而不使用 Smith-Waterman 擴展。
因為提前停止動態編程以生成種子,全局最佳對齊可能包含多個種子,實際上這往往是長對齊的情況。通常對於 1 kb 比對,將有 10-20 個種子。下面將利用這一觀察結果啟發式地加快搜索速度。
BWT-SW 部署了類似的策略,在序列和前綴嘗試之間執行動態編程以查找種子匹配,然後是 Smith-Waterman 擴展。與的算法的主要區別在於,無論 SA 間隔大小如何,一旦分數足夠高,BWT-SW 就會啟動 Smith-Waterman 對齊。有時,重複序列可能與人類基因組中的數千個位置匹配,每次擴展部分匹配可能很慢。
啟發式加速
[編輯]Z-最佳策略
[編輯]到目前為止描述的算法是精確的,因為它能夠提供與 Smith-Waterman 算法相同的結果。雖然在給定長參考序列的情況下,它比標準算法快得多,但對於比對大規模測序數據還不夠快。更仔細的調查表明,即使對於唯一的 500 bp 查詢序列,𝒯(X) 中的幾百萬個節點也可能與具有正比對分數的查詢匹配。這些節點中的大多數是隨機匹配或在短的低複雜度區域中匹配。參觀所有這些都是浪費。
為了加速對齊,在外循環中遍歷𝒢(W),在內循環中遍歷𝒯(X),並且在𝒢(W) 中的每個節點u 處,只保留𝒯(X) 中匹配的前Z 個最佳得分節點u,而不是保留所有匹配的節點。這種啟發式策略稱為 Z-best。當然,當應用 Z-best 策略時,當錯誤匹配具有更高的分數時,可能會錯過包含在真實對齊中的種子。但是,如果查詢與引用幾乎相同,則這種情況發生的頻率較低。另外,如果真對齊很長並且包含很多種子,那麼所有種子都是假的可能性很小。在模擬和真實數據(結果部分)中,發現即使 Z=1 也適用於高質量的 200 bp 讀數(<5% 測序錯誤率)。將 Z 增加到 10 或更高會略微提高準確性,但會大大降低對齊速度。
為了減少對齊錯誤,除了前向對齊之外,還將反向查詢序列與反向參考序列對齊,即反向反向對齊。理想情況下,正向-正向和反向-反向對齊應該產生相同的結果,但如果真正對齊中的種子具有低分後綴(或前綴),則正向-正向(或反向-反向)對齊可能會錯過它,同時結合兩輪對齊減少機會。此外,如果前向對齊中的最佳對齊包含許多種子匹配,則其為假的可能性也很小。在實現中,如果最佳對齊默認包含 5 個或更多種子,則不應用反向-反向對齊。
在 Smith-Waterman 擴展之前過濾種子
[編輯]與 BLAST 一樣,BLAT 和 SSAHA2 都報告了所有重要的比對或通常是數十個得分最高的比對,但這並不是讀取映射中最需要的輸出。通常對覆蓋查詢序列的每個區域的最佳比對或最好的少數比對更感興趣。例如,假設一個 1000 bp 的查詢序列由來自一條染色體的 900 bp 片段和來自另一條染色體的 100 bp 片段組成; 900 bp 片段中的 400 bp 是高度重複的序列。對於 BLAST,要知道這是一個嵌合讀取,需要要求它報告 400 bp 重複的所有比對,這既昂貴又浪費,因為通常對包含在更長的唯一序列中的短重複序列的比對不感興趣序列。在這個例子中,一個有用的輸出是分別報告 900 bp 和 100 bp 片段的一個比對,並指出這兩個片段是否有可能使最佳比對不可靠的次優比對。這樣的輸出簡化了下游分析並節省了重建重複序列的詳細比對的時間。
在 BWA-SW 中,如果查詢上重疊區域的長度小於較短查詢段長度的一半,就說兩個對齊是不同的。的目標是找到一組不同的對齊方式,使集合中每個對齊方式的分數總和最大化。這個問題可以通過動態編程解決,但在的例子中,讀取通常是完全對齊的,貪婪的近似會很好地工作。在實際實現中,根據對齊分數對局部對齊進行排序,從最好的一個掃描排序列表,如果它與所有保留的分數較大的對齊不同,則保留一個對齊;如果對齊 a2 被拒絕,因為它與 a1 沒有區別,認為 a2 是 a1 的次優對齊,並使用此信息來近似映射質量。
因為只保留在查詢序列上基本不重疊的比對,所以不妨丟棄對最終比對沒有貢獻的種子。可以在 Smith-Waterman 擴展之前使用另一種啟發式方法檢測此類種子,從而可以節省花費在不必要擴展上的時間。為了識別這些種子,將包含在條帶中的種子連結起來(默認條頻寬度為 50 bp)。如果在查詢序列上,一條短鏈完全包含在一條長鏈中,並且短鏈中的種子數低於長鏈中種子數的十分之一,將丟棄短鏈中的所有種子,基於觀察到在這種情況下,短鏈很少能比長鏈產生更好的對齊。與 Z-best 策略不同,這種啟發式對對齊精度沒有明顯影響。在 1000 個 10 kb 模擬數據上,它將運行時間減半,而不會降低精度。
近似映射質量
[編輯]Li,H。 等。 (2008) 引入了映射質量的概念來估計查詢序列被放置在錯誤位置的機率。 如果對齊算法保證找到所有局部對齊,則映射質量僅由這些局部對齊決定。 然而,隨著 BWA-SW 部署啟發式規則,產生錯誤對齊的機會也與啟發式有關。 為了估計 BWA-SW 比對的映射質量,擬合了一個經驗公式:250·c1·c2·(S1−S2)/S1,其中 S1 是最佳比對的得分,S2 是次優比對的得分 ,如果對齊覆蓋超過四個種子,則 c1 等於 1,否則為 0.5,如果通過正向-正向和反向-反向對齊找到最佳對齊,則 c2 等於 1,否則為 0.2。
結果
[編輯]簡介
[編輯]BWA-SW 算法是作為 BWA 程序(Li 和 Durbin,2009)的一個組件實現的,該程序在 GNU 通用公共許可證 (GPL) 下分發。該實現將 BWA 索引和查詢 FASTA 或 FASTQ 文件作為輸入,並以 SAM 格式輸出對齊(Li et al., 2009)。查詢文件通常包含許多序列(讀取)。依次處理每個查詢序列,如果適用,使用多個執行緒。內存使用由 FM-index 主導,人類基因組約為 3.7 GB。每個查詢所需的內存大致與序列長度成正比。在典型的測序讀取中,總內存小於 4 GB;在一個具有 100 萬個鹼基對 (Mbp) 的查詢序列上,峰值內存總共為 6.4 GB。
在實現中,嘗試根據讀取長度和測序錯誤率自動調整參數,使默認設置適用於不同特徵的輸入。這種行為對於不熟悉算法的用戶來說很方便,並且有助於提高讀取長度和錯誤率混合的性能。
模擬數據評價
[編輯]在模擬數據上,從比對中知道正確的染色體坐標,並且評估很簡單。
整體表現
[編輯]表 1 顯示了給定不同讀取長度和錯誤率的 BLAT(v34)、BWA-SW(版本 0.5.3)和 SSAHA2(版本 2.4)的 CPU 時間、可靠對齊讀取的分數和對齊錯誤率。 除非必要,嘗試使用每個對齊器的默認命令行選項。 根據輸入數據的特徵微調選項可能會產生更好的性能。
| Program | Metrics | 100 bp | 200 bp | 500 bp | 1000 bp | 10 000 bp | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 2% | 5% | 10% | 2% | 5% | 10% | 2% | 5% | 10% | 2% | 5% | 10% | 2% | 5% | 10% | ||
| BLAT | CPU sec | 685 | 577 | 559 | 819 | 538 | 486 | 1078 | 699 | 512 | 1315 | 862 | 599 | 2628 | 1742 | 710 |
| Q20% | 68.7 | 25.5 | 3.0 | 92.0 | 52.9 | 7.8 | 97.1 | 86.3 | 21.4 | 97.7 | 96.4 | 39.0 | 98.4 | 99.0 | 94.0 | |
| errAln% | 0.99 | 2.48 | 5.47 | 0.55 | 1.72 | 4.55 | 0.17 | 1.12 | 4.41 | 0.01 | 0.52 | 3.98 | 0.00 | 0.00 | 1.28 | |
| BWA-SW | CPU sec | 165 | 125 | 84 | 222 | 168 | 118 | 249 | 172 | 152 | 234 | 168 | 150 | 158 | 134 | 120 |
| Q20% | 85.1 | 62.2 | 19.8 | 93.8 | 88.7 | 49.7 | 96.1 | 95.5 | 85.1 | 96.9 | 96.5 | 95.0 | 98.4 | 98.5 | 98.1 | |
| errAln% | 0.01 | 0.05 | 0.17 | 0.00 | 0.02 | 0.13 | 0.00 | 0.00 | 0.04 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | |
| SSAHA2 | CPU sec | 4872 | 7962 | 9345 | 1932 | 2236 | 5252 | 3311 | 8213 | 6863 | 1554 | 1583 | 3113 | – | – | – |
| Q20% | 85.5 | 83.8 | 78.2 | 93.4 | 93.1 | 91.9 | 96.6 | 96.5 | 96.1 | 97.7 | 97.6 | 97.4 | – | – | – | |
| errAln% | 0.00 | 0.01 | 0.19 | 0.01 | 0.00 | 0.01 | 0.00 | 0.01 | 0.04 | 0.00 | 0.00 | 0.00 | – | – | – | |
| 從人類基因組模擬了大約 10 000 000 bp 不同讀長和錯誤率的數據。 20% 的錯誤是從幾何分布(密度:0.7·0.3l−1)繪製的插入缺失長度的插入錯誤。 這些模擬讀數分別使用 BLAT(選項 -fastMap)、BWA-SW 和 SSAHA2(選項 -454,用於 100 和 200 bp 讀數)與人類基因組對齊。 然後將對齊的坐標與模擬坐標進行比較以查找對齊錯誤。 在此表的每個單元格中,三個數字是 Intel E5420 2.5 GHz CPU 單核上的 CPU 秒數、映射質量大於或等於 20 (Q20) 的對齊百分比以及 Q20 對齊中的錯誤對齊百分比 . SSAHA2 和 BWA-SW 報告映射質量; BLAT 映射質量估計為最佳和次佳對齊分數之差除以最佳對齊分數的 250 倍(與 BWA-SW 的計算基本相同)。 | ||||||||||||||||
從表 1 可以看出,BWA-SW 顯然是最快的,在所有輸入上都比 BLAT 和 SSAHA2 快幾倍,並且其速度對讀取長度或錯誤率不敏感。當查詢較長或錯誤率較低時,BWA-SW 的準確率與 SSAHA2 相當。鑑於短且容易出錯的讀取,SSAHA2 更準確,儘管它必須花費更多時間來對齊此類讀取。 SSAHA2 未在 10 kb 讀取上進行測試,因為它最初不是為此任務設計的,因此性能不佳。帶有 -fastMap 選項的 BLAT 比 SSAHA2 快,但精度較低。在默認選項下,BLAT 比 SSAHA2 慢幾到幾十倍。與 -fastMap 模式相比,精度更高,但總體上仍低於 BWA-SW(數據未顯示)。
在內存上,BWA-SW 和 BLAT 都使用 ∼4 GB 內存。 SSAHA2 使用 2.4 GB 使用默認選項進行≥500 bp 的讀取,使用 5.3 GB 使用 -454 選項進行更短的讀取,這會增加哈希表中存儲的種子序列的數量並因此增加內存。此外,BWA-SW 支持多執行緒,因此如果在多核計算機上運行,每個 CPU 核心可能占用更少的內存。 SSAHA2 和 BLAT 目前不支持多執行緒。
首先研究給定嵌合讀數的每個對準器的行為。為此,製作了兩個嵌合讀數,它們都由一個染色體位置的 1000 bp 片段和另一個位置的 300 bp 片段組成。兩次讀取之間的主要區別在於,第二次讀取中的 1000 bp 片段具有~750 bp 的重複序列,而第一次讀取是高度獨特的。當將兩個嵌合讀數與人類基因組進行比對時,BWA-SW 會根據需要報告四種比對,每個片段一個。最新的 SSAHA2 無法在兩個讀取中找到 300 bp 片段的對齊,儘管如果將 300 bp 片段作為單獨讀取對齊,它能夠找到對齊。默認情況下,舊版本 (1.0.9) 能夠在第一次讀取中對齊 300 bp 片段,但對於第二次讀取,需要切換到更徹底但速度更慢的配置,將所有命中報告到 750 bp重複。帶有 -fastMap 的 BLAT 沒有找到第二次讀取的 300 bp 片段的對齊。在這兩個例子中,只有 BWA-SW 有足夠的能力來檢測嵌合體。
此外,BWA-SW 很少產生高質量的假嵌合比對。例如,假設 10 000 1 kb 讀數有 10% 的錯誤但在模擬中沒有嵌合體,BWA-SW 預測 18 個嵌合讀數。這些讀取上錯誤對齊的片段的映射質量僅為 2.4(最大 11),這意味著 BWA-SW 意識到這些嵌合體是不可靠的。正如預期的那樣,由於鹼基錯誤較低,BWA-SW 產生的假嵌合讀數較少。
真實數據評估
[編輯]由於缺乏基本事實,對真實數據的評估變得複雜。然而,仍然可以通過比較兩個對齊器的結果來評估相對準確性,使用真實對齊往往具有相當高的對齊分數的原則,因為大多數錯誤是由於未能找到種子而產生的。
假設使用兩個對齊器 A 和 B 對齊讀取並得到不同的結果。如果 A 和 B 都給出低映射質量,則對齊是不明確的,並且任何對齊是否錯誤都無關緊要。如果 A 的映射質量很高,而 A 的對齊分數比 B 差,則 A 對齊可能是錯誤的;即使A對齊分數比B好一點,A對齊也不可靠,A給出的高映射質量仍然值得懷疑。實際上,定義「稍微好一點」的對齊分數需要對分數差異設置任意閾值,因此這種評估方法是近似的。
表 2 匯總了 454 個讀數,這些讀數僅由一個比對器映射或映射到不同位置,並且被 BWA-SW 或 SSAHA2 指定的映射質量大於或等於 20。可以看到 BWA-SW 往往會錯過高錯誤率的短對齊(其中 946 個),這與對模擬數據的評估一致。 SSAHA2 由於不同的原因錯過了對齊。在 1188 次讀取中,SSAHA2 產生明顯錯誤的比對。通過分配低映射質量意識到這些對齊是錯誤的,但無論如何都錯過了真正的對齊。
| Condition | Count | BWA-SW | SSAHA2 | ||||
|---|---|---|---|---|---|---|---|
| avgLen | avgDiff | avgMapQ | avgLen | avgDiff | avgMapQ | ||
| BWA-SW≥20; SSAHA2 unmapped | 0 | – | – | – | – | – | – |
| BWA-SW≥20 plausible; SSAHA2<20 | 1188 | 398.2 | 1.3% | 178.4 | 198.3 | 13.1% | 3.9 |
| BWA-SW≥20 questionable | 40 | 183.0 | 7.8% | 41.2 | 280.3 | 9.4% | 2.4 |
| SSAHA2≥20; BWA-SW unmapped | 946 | – | – | – | 75.4 | 6.3% | 51.2 |
| SSAHA2≥20 plausible; BWA-SW<20 | 47 | 129.0 | 9.3% | 2.5 | 200.5 | 8.8% | 34.4 |
| SSAHA2≥20 questionable | 185 | 400.2 | 1.7% | 13.4 | 399.2 | 2.9% | 216.4 |
| 從 SRR003161 中統一選擇的總共 137 670 454 個讀數分別用 BWA-SW 和 SSAHA2 映射到人類基因組。如果 BWA-SW 和 SSAHA2 對齊的最左側坐標相差超過 355 bp(平均讀取長度),則稱讀取不一致對齊。為每個比對計算一個分數,該分數等於匹配數減去三乘以比對區域中的差異(錯配和缺口)數。如果從 BWA-SW 比對得出的分數減去從相同讀取的 SSAHA2 比對得出的分數大於或等於 20(即 BWA-SW 比對足夠好),則認為 BWA-SW 比對是合理的;否則,據說 BWA-SW 對齊是有問題的。合理的和有問題的 SSAHA2 比對以類似的方式定義。表中,『BWA-SW≥20』表示映射質量高於20的BWA-SW比對。BWA-SW總共遺漏了993(=946+47)個比對,其中SSAHA2比對好,而SSAHA2比對好1188; 40 個 BWA-SW Q20 對齊和 185 個 SSAHA2 Q20 對齊可能是錯誤的。 | |||||||
對於這兩個比對器,大多數錯誤對齊是由於忽略了與最佳報告對齊具有相似分數的對齊。例如,SSAHA2 將讀取 SRR003161.1261578 與映射質量為 244 的 X 染色體對齊,而 BWA-SW 將其與具有相同對齊長度和編輯距離的 2 號染色體對齊。兩個最佳評分比對的存在意味著無法唯一放置讀取,並且高達 244 的映射質量是不准確的。 SSAHA2 提供這種高映射質量可能是因為它忽略了染色體 2 上的匹配。在這個特定示例中,BWA-SW 正確地給出了一個映射質量為零,儘管它可能會忽略其他示例中的替代匹配。
在模擬的 100 和 200 bp 讀數上,帶有 -454 選項的 SSAHA2 比 BWA-SW 提供更好的比對。在這個真實的數據集上,BWA-SW 更準確,可能是因為平均讀取長度相對較長(355 bp)。為了證實這一推測,比較了來自運行 SRR002644 的 99 958 個讀數的兩個對齊器,平均讀數長度為 206 bp。這次 BWA-SW 錯過了 1092 個 SSAHA2 Q20 比對,並產生了 39 個有問題的比對; SSAHA2 漏掉了 325 個,產生了 10 個有問題的。 SSAHA2 在這個較短的數據集上更準確,儘管它比 BWA-SW 慢 9 倍,並且使用的內存多 40%。
討論
[編輯]BWA-SW 是一種有效的算法,用於將幾百個或更多鹼基對的查詢序列與長參考基因組進行比對。對於長查詢或低錯誤率的查詢,其敏感性和特異性往往更高,並且在此類查詢序列上,BWA-SW 的準確性可與迄今為止最準確的對齊器相媲美。此外,BWA-SW 能夠檢測可能由結構變化或參考錯誤組裝引起的嵌合體,這可能對 BLAT 和 SSAHA2 構成挑戰。
BWA-SW、BLAT 和 SSAHA2 都遵循種子和擴展範式。主要區別來自播種策略。 BLAT 和 SSAHA2 將短精確匹配識別為種子,通常長度為 11 或 12 bp。對於分別在長度為 L 和 l 的兩個序列之間的 k-mer 種子,種子的預期數量為 L·l/4k,或者是 105 的數量級,用於與人類基因組的比對。用 Smith-Waterman 算法擴展這些種子是很昂貴的。為了減少不必要的種子擴展,BLAT 和 SSAHA2 默認都使用不重疊的種子,並且需要多個種子匹配,這對於隨機序列應該可以很好地工作,但仍然涉及高度重複區域中的許多種子擴展。 BWA-SW 通過在獨特區域使用一些長間隙種子解決了這個問題。在真實的生物數據上,它節省了許多不必要的種子擴展,並帶來了更好的整體性能。然而,為了減少識別長種子的時間,BWA-SW 只保留了動態規劃矩陣的一小部分,這可能會錯過所有真正匹配的種子。這種啟發式是對齊錯誤的主要來源,特別是對於短查詢,當要對齊的序列之間只有很少的有效唯一種子時。幸運的是,在長對齊上,丟失所有種子的可能性很小。已經證明 BWA-SW 與 SSAHA2 一樣有效。
BWA-SW 在幾個方面與 BWT-SW 不同。首先,BWT-SW 保證找到所有本地匹配,而 BWA-SW 是一種啟發式算法,它可能會錯過真正的命中,但速度要快得多。其次,BWA-SW 對齊兩個 FM 索引,而 BWT-SW 對齊一個序列和一個 FM 索引。為查詢序列構建前綴 DAWG 可能有助於避免在查詢中重複對齊相同的子字符串,從而提高理論時間複雜度。第三,BWA-SW在內循環中遍歷參考前綴trie,而BWT-SW在內循環中遍歷查詢序列。如果沒有啟發式方法,BWA-SW 方法會損害性能,因為必須在遍歷引用前綴樹時用速度換取內存,而在外循環中遍歷它會更有效。儘管如此,應用 Z-best 策略需要知道與查詢子串匹配的得分最高的參考節點,而無需完成動態規劃,因此僅在內部循環中遍歷參考時才有效。第四,BWA-SW 只報告與查詢序列基本不重疊的比對,而 BWT-SW 與 BLAST 一樣,報告所有具有統計意義的比對。 BWA-SW 保留了對齊的關鍵信息,並生成更小更方便的輸出。對於 BWT-SW,最終用戶通常需要對結果進行後處理,以過濾掉許多他們不感興趣的對齊方式。總而言之,鑑於大規模真實數據,BWA-SW 被調整為具有實際實用性。
BWA-SW 的高速主要來自兩種策略:使用 FM 索引和抑制包含在更好匹配中的短重複匹配。雖然第一種策略不適用於基於哈希表的算法,例如 SSAHA2 和 BLAT,但第二種策略可以在此類程序中實現,並且可以通過節省大量構建重複對齊的時間來顯著加速它們。雖然 BWT 的使用減少了重複中不必要的對齊,但與哈希表查找相比,每個 BWT 操作都帶有一個很大的常量。如果基於哈希表的算法結合了其中的一些功能,那麼它們仍有可能比 BWA-SW 更快。
參考文獻
[編輯]- Altschul SF, et al. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Res. 1997;25:3389–3402. [PMC free article] [PubMed] [Google Scholar]
- Blumer A, et al. The smallest automaton recognizing the subwords of a text. Theor. Comput. Sci. 1985;40:31–55. [Google Scholar]
- Burrows M, Wheeler DJ. Technical report 124. Palo Alto, CA: Digital Equipment Corporation; 1994. A block-sorting lossless data compression algorithm. [Google Scholar]
- Campagna D, et al. PASS: a program to align short sequences. Bioinformatics. 2009;25:967–968. [PubMed] [Google Scholar]
- Eaves HL, Gao Y. MOM: maximum oligonucleotide mapping. Bioinformatics. 2009;25:969–970. [PubMed] [Google Scholar]
- Eid J, et al. Real-time DNA sequencing from single polymerase molecules. Science. 2009;323:133–138. [PubMed] [Google Scholar]
- Ferragina P, Manzini G. Proceedings of the 41st Symposium on Foundations of Computer Science (FOCS 2000) IEEE Computer Society; 2000. Opportunistic data structures with applications; pp. 390–398. [Google Scholar]
- Ferragina P, Manzini G. Proceedings of the 41st Symposium on Foundations of Computer Science (FOCS 2000) Redondo Beach, CA, USA: 2000. Opportunistic data structures with applications; pp. 390–398. [Google Scholar]
- Grossi R, Vitter JS. Proceedings on 32nd Annual ACM Symposium on Theory of Computing (STOC 2000) ACM; 2000. Compressed suffix arrays and suffix trees with applications to text indexing and string matching; pp. 397–406. [Google Scholar]
- Hon W.-K, et al. A space and time efficient algorithm for constructing compressed suffix arrays. Algorithmica. 2007;48:23–36. [Google Scholar]
- Jiang H, Wong WH. SeqMap: mapping massive amount of oligonucleotides to the genome. Bioinformatics. 2008;24:2395–2396. [PMC free article] [PubMed] [Google Scholar]
- Jung Kim Y, et al. ProbeMatch: a tool for aligning oligonucleotide sequences. Bioinformatics. 2009;25:1424–1425. [PMC free article] [PubMed] [Google Scholar]
- Kent WJ. BLAT–the BLAST-like alignment tool. Genome Res. 2002;12:656–664. [PMC free article] [PubMed] [Google Scholar]
- Lam TW, et al. Compressed indexing and local alignment of DNA. Bioinformatics. 2008;24:791–797. [PubMed] [Google Scholar]
- Langmead B, et al. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 2009;10:R25. [PMC free article] [PubMed] [Google Scholar]
- Lin H, et al. ZOOM! Zillions of oligos mapped. Bioinformatics. 2008;24:2431–2437. [PMC free article] [PubMed] [Google Scholar]
- Lippert RA. Space-efficient whole genome comparisons with Burrows-Wheeler transforms. J. Comput. Biol. 2005;12:407–415. [PubMed] [Google Scholar]
- Li H, et al. Mapping short DNA sequencing reads and calling variants using mapping quality scores. Genome Res. 2008a;18:1851–1858. [PMC free article] [PubMed] [Google Scholar]
- Li R, et al. SOAP: short oligonucleotide alignment program. Bioinformatics. 2008b;24:713–714. [PubMed] [Google Scholar]
- Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics. 2009;25:1754–1760. [PMC free article] [PubMed] [Google Scholar]
- Li H, et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 2009;25:2078–2079. [PMC free article] [PubMed] [Google Scholar]
- Ma B, et al. PatternHunter: faster and more sensitive homology search. Bioinformatics. 2002;18:440–445. [PubMed] [Google Scholar]
- Malhis N, et al. Slider–maximum use of probability information for alignment of short sequence reads and SNP detection. Bioinformatics. 2009;25:6–13. [PMC free article] [PubMed] [Google Scholar]
- Meek C, et al. Proceedings of 29th International Conference on Very Large Data Bases (VLDB 2003) Berlin, Germany: 2003. OASIS: an online and accurate technique for local-alignment searches on biological sequences; pp. 910–921. [Google Scholar]
- Morgulis A, et al. Database indexing for production megablast searches. Bioinformatics. 2008;24:1757–1764. [PMC free article] [PubMed] [Google Scholar]
- Ning Z, et al. SSAHA: a fast search method for large DNA databases. Genome Res. 2001;11:1725–1729. [PMC free article] [PubMed] [Google Scholar]
- Pearson WR, Lipman DJ. Improved tools for biological sequence comparison. Proc. Natl Acad. Sci. USA. 1988;85:2444–2448. [PMC free article] [PubMed] [Google Scholar]
- Rumble SM, et al. SHRiMP: accurate mapping of short color-space reads. PLoS Comput. Biol. 2009;5:e1000386. [PMC free article] [PubMed] [Google Scholar]
- Schatz M. Cloudburst: highly sensitive read mapping with mapreduce. Bioinformatics. 2009;25:1363–1369. [PMC free article] [PubMed] [Google Scholar]
- Smith AD, et al. Using quality scores and longer reads improves accuracy of Solexa read mapping. BMC Bioinformatics. 2008;9:128. [PMC free article] [PubMed] [Google Scholar]
- Weese D, et al. RazerS–fast read mapping with sensitivity control. Genome Res. 2009;19:1646–1654. [PMC free article] [PubMed] [Google Scholar]
- Zhang Z, et al. A greedy algorithm for aligning DNA sequences. J. Comput. Biol. 2000;7:203–214. [PubMed] [Google Scholar]




![{\textstyle [{\underline {R}}(W),{\overline {R}}(W)]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/0c7e276a085f0472a7e4a0d8b5841a87182b5027)








![{\displaystyle 4n[log_{2}n]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/21b7ee22a999d3f3f7c016da5e118521a61fe2a5)
![{\displaystyle [log_{2}n]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/eeb4f66d3a7cc7807792dbae6d1bc85071c6ffe7)
![{\displaystyle 2n+n[log_{2}n]/32}](https://wikimedia.org/api/rest_v1/media/math/render/svg/3ea8f5f4e475ee0a762bb78f020d55963c2ed575)



![{\displaystyle {\psi ^{-1}}(i)=C(B[i]+O(B[i],i)}](https://wikimedia.org/api/rest_v1/media/math/render/svg/498417e801535c91fa09c38e24a9f543bf1ea1ff)


![{\displaystyle 4n+n[log_{2}n]/8}](https://wikimedia.org/api/rest_v1/media/math/render/svg/8ffa698a0aa4c216afce50a36e5bc4da0bf49f5c)



![{\displaystyle \textstyle \sum _{i=1}^{l+1}\displaystyle q^{\prime }\cdot (1-\delta _{{\hat {b}}_{i},b_{i}})+\textstyle \sum _{i=1}^{l}\displaystyle q_{i}[1-\delta _{{\hat {c}}_{i},g({\hat {b}}_{i},{\hat {b}}_{i+1})}]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/cf45c6186cecd77280cab632976f77ba7b894f25)





![{\displaystyle f_{i+1}({\hat {b}}_{i+1})=min_{{\hat {b}}_{i}}\left\{f_{i}({\hat {b}}_{i})+q^{\prime }\cdot (1-\delta _{{\hat {b}}_{i+1},b_{i+1}})+q_{i}\cdot [1-\delta _{c_{i},g({\hat {b}}_{i},{\hat {b}}_{i+1})}]\right\}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/0a27e8e941e4ccf9de34aa1a6bedd3532ee73e25)









